Questions d’archéométrie : l’analyse de l’ADN ancien en archéologie
par Diane Martin-Moya. Doctorante en bioarchéologie à l’Université de Montréal et l’Université du Québec à Trois-Rivières
À quoi sert l’ADN ancien en archéologie et que peut-on en apprendre ?
Le développement des analyses d’ADN ancien constitue une véritable révolution en bioarchéologie. Cette nouvelle discipline intégrant un nouveau jeu de données biologiques inédites a contribué à l’avancement des connaissances sur un large spectre de sujets, dont voici quelques exemples majeurs.
Identifier des membres de la lignée humaine comme Denisova simplement à partir d’une molaire et d’une phalange découverte dans une grotte de l’Altaï en Sibérie datée de plus de 41 000 ans. Les Denisoviens sont les premiers à avoir été identifiés grâce à l’ADN ancien. Comme les hommes de Néandertal, les Denisoviens ont contribué au patrimoine génétique des hommes anatomiquement modernes et surtout ceux issus de l’Asie du Sud-Est.
Reconstruire à l’échelle biologique l’histoire des populations en suivant les migrations et dispersions des espèces tout autour du globe. L’ADN ancien a permis d’identifier plusieurs épisodes d’échanges en Europe avec des populations en provenance du Proche et Moyen-Orient il y a plus de 8 000 ans. Cet apport n’était cependant pas homogène et plusieurs groupes de chasseurs-cueilleurs en Scandinavie en sont restés isolés. La majorité du patrimoine génétique européen moderne est issue de cinq zones d’influence à l’Âge du Bronze.
Reconstruire les processus de domestication. Le Néolithique connaît les premières formes de spéciation de certaines espèces animales et végétales et des changements dans les pratiques alimentaires, dont la consommation du lait. L’ADN ancien démontre que la digestion du lait est l’adaptation la plus massivement sélectionnée dans l’histoire de l’évolution chez les populations eurasiennes. Les populations ne consommant pas ou peu de lait de vache après l’âge du sevrage ne produisent plus la molécule permettant sa digestion. C’est ce qui explique la persistance chez certaines populations de l’intolérance au lactose.
Identifier des espèces éteintes. En 2021, le plus vieil ADN fossile jamais séquencé vieux de 1,2 million d’années a permis de mieux comprendre l’histoire d’une espèce éteinte, les mammouths. Trois espèces différentes ont existé au cours du Pléistocène, dont une espèce ancestrale qui a traversé l’Eurasie pour atteindre l’Amérique du Nord. Grâce à l’ADN ancien, nous savons que son descendant, le mammouth laineux, disparu il y a environ 4 000 ans, démontrait une meilleure adaptation aux températures froides.
Explorer le développement et laproliférationde virus ou l’hygiène et la santé des populations passées. L’introduction d’agents pathogènes (variole, typhus, grippe, et autres) en Amérique durant la colonisation a eu pour conséquence de décimer plus de 90 % des populations autochtones. L’ADN ancien a permis d’évaluer l’impact de ce goulot d’étranglement dévastateur sur la diversité du patrimoine génétique des Premières Nations il y a 500 ans.
Respecter le souhait des populations descendantes en n’altérant pas les corps des défunts tout en permettant d’en connaître plus sur leurs ancêtres. Afin de limiter l’altération et la manipulation des restes humains, des archéogénéticiens ont contribué à développer des méthodes d’échantillonnage alternatives en récupérant le tartre dentaire. Le tartre constitue une ressource importante d’informations permettant de renseigner sur l’hygiène, l’alimentation, les pathologies et même les mouvements de populations.
Depuis les années 2000, les archéogénéticiens ont généré des millions de séquences d’ADN permettant depuis ces dernières années de passer de l’analyse d’un génome isolé à celui de génomes à l’échelle d’une population, et ce, même pour les populations éteintes il y a des milliers d’années.
Où trouve-t-on l’ADN ancien ?
N’importe quel organisme possédant un ADN peut passer sous la loupe de l’archéogénéticien : la faune, la flore et même le microbiome. On peut le trouver directement dans les os, les cheveux, les poux, le tartre sur les dents, les coprolithes, une ancienne gomme à mâcher ou un grain de maïs préservé. Il est même possible de tout simplement récupérer un échantillon de sol archéologique, car ils regorgent de microorganismes.
Qu’est-ce qui caractérise l’ADN ?
L’ADN (ou acide désoxyribonucléique) est une macromolécule présente dans chaque cellule de chaque être vivant. Il contient l’information génétique, appelée génotype, qui est présente dans le génome. Le génome peut être comparé à un manuel dont l’alphabet (son unité de base) se compose de quatre nucléotides : Adénine, Cytosine, Thymine et Guanine (ACTG). L’enchaînement successif des nucléotides est réparti dans les gènes et constitue la séquence d’ADN.
La génétique, comme beaucoup de disciplines, consiste à observer les différences et les similarités à l’échelle moléculaire. Des différenciations que l’on appelle « mutations », et qui sont au niveau des nucléotides, s’accumulent au fil du temps. Ce sont ces différences et ces similarités qui permettent d’établir les relations phylogénétiques entre tous les organismes du vivant.
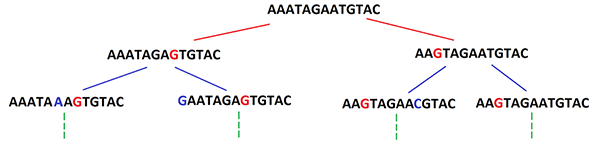
Il est même possible de retracer la lignée des espèces actuellement éteintes. Tous les organismes sont reliés par un ancêtre commun avec lequel ils partagent une partie de l’information génétique. L’ADN peut être utilisé pour (i) estimer les périodes de divergence dans le temps et (ii) remonter dans l’échelle de l’évolution pour reconstruire « l’arbre du vivant ». Il existe de nombreuses variables pour expliquer ces divergences dans le temps. Les principales forces évolutives au travers des mutations sont la sélection naturelle, la dérive génétique et les flux géniques entre populations.
Et à l’échelle humaine ?
Chaque individu hérite de la moitié du patrimoine génétique du père et de la moitié de celui de la mère. Ainsi, cet individu transmettra par la suite seulement un quart des gènes de chacun de ses parents, et ainsi de suite au fil des générations. Ce sont ces mutations accumulées au cours du temps qui permettent de reconstruire à l’échelle locale l’histoire génétique des populations anciennes. Avant les grandes avancées méthodologiques en archéogénétique, les analyses ciblaient surtout l’ADN mitochondrial (présent dans les mitochondries dans chaque cellule) transmis seulement de la mère aux enfants.
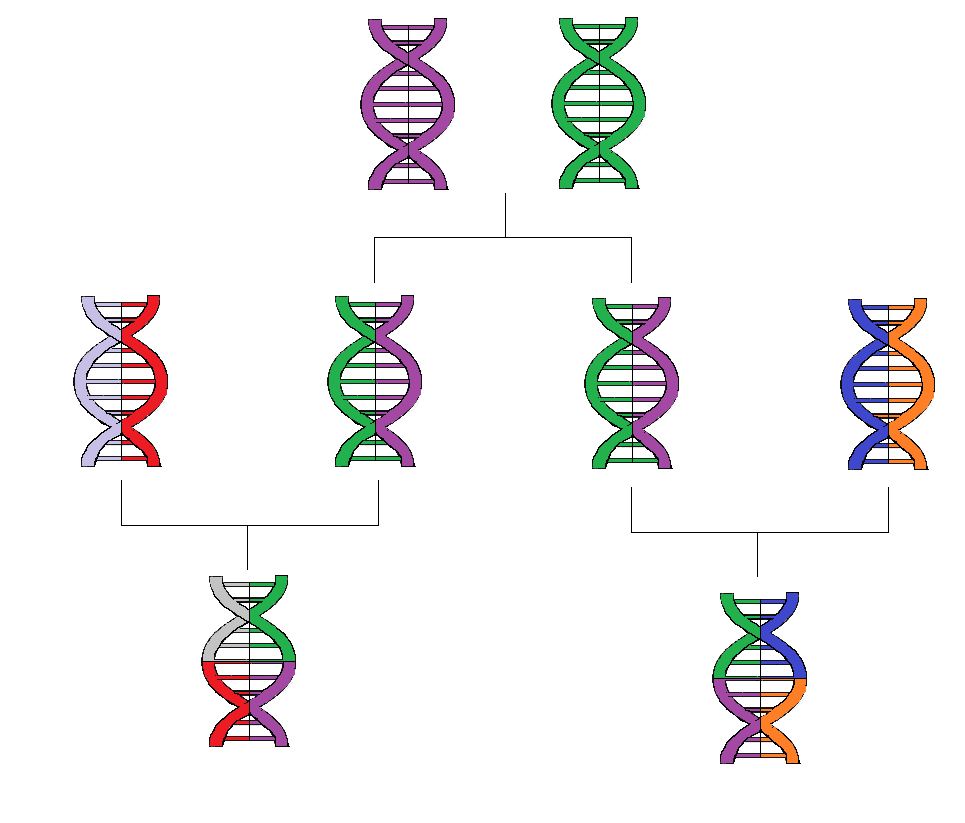
Qu’est-ce qui caractérise l’ADN ancien ?
Après la mort de l’organisme, l’ADN perd graduellement, au fil du temps, ses fragments contenant l’information génétique. L’ADN possède une structure moléculaire très fragile et centrale aux processus évolutifs pour générer les mutations. Toutefois, la mort de l’organisme rend l’ADN sensible aux facteurs environnementaux et aux processus de contamination. Le temps et l’environnement sont donc des facteurs clés pour déterminer la qualité de l’ADN ancien. Néanmoins, l’environnement peut aussi être bénéfique. La fonte du pergélisol, qui constitue une barrière aux facteurs extérieurs, a permis ces dernières années de faire des découvertes majeures sur d’anciennes espèces parfaitement préservées. Au Yukon, une momie d’un bébé loup vieux de 57 000 ans a été découverte dans le pergélisol. La fonte du pergélisol a révélé aussi des lionceaux et ours des cavernes en Sibérie.

Comment analyse-t-on l’ADN ancien ?
L’ADN dégradé ne possède que quelques fragments de sa séquence d’origine, ce qui entraîne des coûts élevés liés à son analyse. Il est donc important de bien cerner la question de recherche et de préparer l’échantillonnage en fonction de celle-ci. Par exemple, dans le cas de l’analyse d’un cimetière au Québec ayant pour objectif de reconstruire l’histoire de la population ou des agents pathogènes, nous allons sélectionner préférentiellement un os (dent ou partie du crâne). À plus large échelle, si nous voulons reconstruire un environnement passé, un échantillon de sol permet d’accéder à toute l’information génétique des organismes présents (insectes, flore, faune, champignons et peut-être même de l’humain).
I. Sur le terrain
L’échantillonnage peut se faire directement sur le terrain ou à partir de collections muséales sur les restes ostéologiques, les sols ou les restes organiques. Il est important de minimiser la contamination exogène pour maximiser les chances de trouver de l’ADN ancien.
II. En laboratoire
Chaque échantillon est exposé à son environnement de découverte qui regorge de matériaux contaminants. Il est donc important de minimiser les sources potentielles de contamination afin d’éviter de séquencer de l’ADN exogène (comme celui du fouilleur par exemple). L’archéogénéticien est donc soumis à un environnement et à un équipement de travail très règlementé pour minimiser la contamination. Les extractions et les analyses se font dans des salles blanches où l’accès et les déplacements sont tous régulés et les changements d’équipements fréquents.



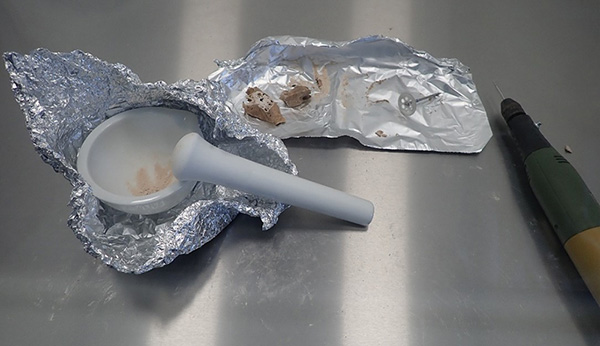
Extraire l’ADN > Isoler l’ADN > Préparer l’ADN
Très simplement dit, il y a trois grandes étapes en laboratoire pour transformer l’échantillon extrait du sol à l’état d’objet d’étude intelligible pour l’archéogénéticien. La première consiste à extraire la portion la moins exposée aux agents extérieurs et à la purifier pour isoler l’information génétique. Deuxièmement, il faut compiler cette information génétique dans une « librairie ». La librairie est donc constituée de l’enchaînement de plusieurs lettres, mais les mots sont cryptiques. Pour la dernière phase, il faut indexer cette librairie pour ses passages dans le séquenceur. Pour s’assurer de ne pas perdre de l’information, il faut en créer des copies par amplification à l’aide d’une réaction en chaîne de la polymérase (communément désignée PCR pour Polymerase Chain Reaction). Pour finir, le séquençage permettra de référencer les nucléotides dans l’ordre d’apparition exacte dans la séquence d’ADN pour la lire et nous permettre de passer à l’étape de l’analyse.
III. Derrière l’ordinateur
Il ne reste plus qu’à lire et nettoyer la séquence ! La lecture se fait en croisant la séquence aux bases de données disponibles pour (i) identifier quelles portions de la séquence d’ADN ont été récupérées, (ii) déterminer si elle est perturbée par les pertes et recombinaisons géniques dues aux temps et aux facteurs externes, (iii) comparer cette séquence aux bases de données pour nettoyer des fragments d’ADN exogène, et (iv) extraire les informations présentes. En fonction de la question de recherche, cette étape pourra mener à l’identification d’ancêtres communs qui permettront de reconstruire l’arbre phylogénétique ou généalogique, à l’identification de pathologies ou, dans le cadre d’études environnementales, à l’identification des diverses espèces fauniques, botaniques, fongiques, bactériennes, etc. ayant fait l’objet d’un séquençage.
Une discipline qui avance au rythme de l’optimisation technologique et technique
L’archéogénétique ne cesse d’évoluer et de perfectionner ses méthodes d’analyses et de réduction de la contamination.
Pendant longtemps les archéogénéticiens étaient dépendants d’une importante quantité de restes osseux et ne pouvaient séquencer au-delà de 15 000 ans. Il leur est maintenant possible de remonter à 1,2 million d’années et d’extraire de l’ADN ancien à partir de gommes à mâcher, de coprolithes, de tartre dentaire et de résidus salivaires. Plusieurs chercheurs s’affairent également à mieux comprendre les processus de transformation de la séquence d’ADN d’origine après la mort de l’organisme pour récolter une plus grande proportion de la séquence d’ADN fragmenté, voire même de la reconstruire.
Comme dans toute étude archéologique, il y a une importante part d’indétermination et d’interprétation ; on travaille avec des informations parcellaires pour retracer l’histoire à l’échelle d’une population. L’archéogénétique ne fait pas exception, mais grâce à l’accumulation de nouvelles données sur des périodes ou des populations particulières, il est désormais possible de combler certains fragments « inconnus » et d’ainsi reformuler ou améliorer nos interprétations.
Et au Québec ?
Grâce à l’amélioration des banques de données généalogiques couplées à l’archéogénétique, les populations de référence ne cessent de s’affiner. Au Québec, il existe une banque de données de référence généalogique qui couvre la période de 1621 à 2008, avec plus de 5 millions d’individus/ancêtres identifiés (BALSAC : http://balsac.uqac.ca/). Ces banques de données permettent de réviser nos connaissances sur l’histoire démographique et sociale des populations émergentes en Nouvelle-France ; jusqu’à identifier les noms des colons aux débuts des stratégies de peuplements, en croisant des données générales et locales sur plusieurs générations.
Depuis 2020, le premier laboratoire TRACE (Criminalistique et ADN ancien) est en cours de développement à l’Université du Québec à Trois-Rivières. Ce laboratoire permet d’ouvrir de nouveaux champs d’études, de formations et de réflexions in situ à la pratique archéologique québécoise. Trois axes de recherches en ADN ancien sont à ce jour explorés au laboratoire : (i) la composition des populations de l’époque coloniale à Montréal à travers deux cimetières majeurs, celui de Notre-Dame (1691-1796) et celui de Saint-Antoine (1799-1855); (ii) la composition des agents pathogènes et des grandes épidémies au début de la colonisation à Montréal ; et (iii) l’identification des naufragés du bateau Irlandais Carricks, qui a sombré en 1847 et a été découvert dans des fosses sur la plage de Cap-des-Rosiers.
Glossaire
Biologie cellulaire et moléculaire : Chaine vidéo par Jonathan Dumas : https://youtube.com/playlist?list=PL-vrUYMihOdXeOG6bivBEMYlnOyKwnYUo
Spéciation : « Processus évolutif par lequel de nouvelles espèces vivantes se forment à partir d’ancêtres communs. Les espèces s’individualisent à partir de populations appartenant à une espèce » d’origine (sauf exception pour certains végétaux) ».
consulté le 25-02-2022 - https://fr.wikipedia.org/wiki/Sp%C3%A9ciation)
Adaptation : « Transformation d’un organisme pour maximiser ses capacités de survie et de reproduction face aux pressions externes et/ou internes ».
consulté le 25-02-2022 - https://www.larousse.fr/dictionnaires/francais/adaptation/1003)
Domestication : « la domestication d’une espèce, animale ou végétale, est l’acquisition, la perte ou le développement de caractères morphologiques, physiologiques ou comportementaux nouveaux et héréditaires, résultant d’une interaction prolongée, d’un contrôle voire d’une sélection délibérée de la part des communautés humaines.»
consulté le 25 – 02 – 2022 : https://fr.wikipedia.org/wiki/Domestication)
Plus à voir et à lire :
TED Talk par Svante Pääbo (paléogénéticien au Max Planck Institute – Leipzik, Allemagne): https://www.ted.com/talks/svante_paabo_dna_clues_to_our_inner_neanderthal
Reportages sur les recherches en ADN ancien au Québec (Université du Québec à Trois-Rivières, QC, Canada)
Paléogénomique et génétique moléculaire humaine au Musée de l’Homme à Paris (France)
Fiche explicative & médiathèque sur la paléogénomique par l’INRAP (France)
Émission de radio sur l’ADN et couleurs des cheveux chez les néandertaliens
Conférence sur la peste :
https://www.inrap.fr/des-souches-divergentes-de-la-peste-il-y-5-000-ans-en-eurasie-11699
Articles du Smithsonian Magazine sur les découvertes du pergélisol
Références :
Brown, S., Higham, T., Slon, V., Pääbo, S., Meyer, M., Douka, K., ... & Buckley, M. (2016). Identification of a new hominin bone from Denisova Cave, Siberia using collagen fingerprinting and mitochondrial DNA analysis. Scientific reports, 6 (1), 1-8.
Dabney, J., Knapp, M., Glocke, I., Gansauge, M. T., Weihmann, A., Nickel, B., ... & Meyer, M. (2013). Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments. Proceedings of the National Academy of Sciences, 110 (39), 15758-15763.
Harbeck, M., Seifert, L., Hänsch, S., Wagner, D. M., Birdsell, D., Parise, K. L., ... & Scholz, H. C. (2013). Yersinia pestis DNA from skeletal remains from the 6th century AD reveals insights into Justinianic Plague. PLoS pathogens, 9 (5), e1003349.
Harding, T., Milot, E., Moreau, C., Lefebvre, J. F., Bournival, J. S., Vézina, H., ... & Labuda, D. (2020). Historical human remains identification through maternal and paternal genetic signatures in a founder population with extensive genealogical record. American journal of physical anthropology, 171 (4), 645-658.
Lalueza-Fox C, Römpler H, Caramelli D, Stäubert C, Catalano G, Hughes D, Rohland N, Pilli E, Longo L, Condemi S, de la Rasilla M, Fortea J, Rosas A, Stoneking M, Schöneberg T, Bertranpetit J, Hofreiter M : A melanocortin 1 receptor allele suggests varying pigmentation among Neanderthals. Science. 2007, 318 : 1453-1455. 101,126/science.1147417.
van der Valk, T., Pečnerová, P., Díez-del-Molino, D., Bergström, A., Oppenheimer, J., Hartmann, S., ... & Dalén, L. (2021). Million-year-old DNA sheds light on the genomic history of mammoths. Nature, 591 (7849), 265-269.
Lazaridis, I., Patterson, N., Mittnik, A., Renaud, G., Mallick, S., Kirsanow, K., ... & Berger, B. (2014). Ancient human genomes suggest three ancestral populations for present-day Europeans. Nature, 513 (7518), 409.
Ségurel, L., & Bon, C. (2017). On the evolution of lactase persistence in humans. Annual review of genomics and human genetics, 18, 297-319.